背景[1-7]
qPCR Master Mix(qPCR预混液Mix)是2×用于实时定量PCR的预混合溶液,Mix中含有HotStart DNA Polymerase、SYBR Green I、dNTPs、Mg2+以及优化的缓冲体系。仅需加入待测模板和引物即可进行实时荧光定量PCR。qPCR Master Mix
提供了一个简便、灵敏、可靠地进行实时荧光PCR检测的完整系统。产品包含了经过优化的缓冲液组分、dNTP、化学修饰的热启动DNA聚合酶和MgCl2溶液及EvaGreen染料混合物,可用于以未标记引物进行的掺入法Real Time定量PCR。
EvaGreen是一种用于实时定量PCR(qPCR)的DNA结合染料。该染料的诸多优点使它远胜于SYBR Green I。除了有相似的光谱特性,EvaGreen有三个主要特点使它区别于SYBR Green I。首先,EvaGreen对PCR的抑制性远小于SYBR Green I。因此,使用EvaGreen进行的qPCR实验可以使用快速PCR步骤。同时,EvaGreen在实验中可以使用较高的浓度,从而获得远强于SYBR Green I扩增信号。较高浓度的EvaGreen也消除了“染料重分布”的缺陷,使EvaGreen既可用于多重PCR,也可用于高分辨率(高清晰)熔解曲线分析(HRM)。
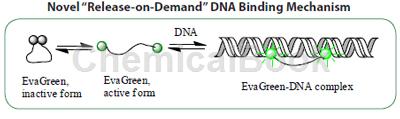
该分析正被越来越多的用于PCR后的基因分型和异源双链分析。由于SYBR Green I对PCR的抑制性,从而要求其使用浓度必须很低,因此SYBR Green I无法解决由低浓度造成的染料重分布问题,既不能用于多重PCR也不能用于HRM。同时,染料重分布问题也可能影响常规熔解曲线的可靠性,因为低熔点的DNA链可能由于这种原因而无法检测到。
第二,EvaGreen的稳定性极好。在正常的储存、操作和PCR过程中不会被破坏。在缓冲溶液中的染料可以安全的储存在室温或冰箱里,也可以反复冻融。与之相反,SYBR Green I不稳定而且降解后对PCR抑制性更强。EvaGreen降低了细胞膜透性,因而比SYBR Green 1更加安全。独立实验室的测试结果显示,EvaGreen既没有诱变性也没有细胞毒性。相反,虽然SYBR Green I本身诱变性很弱,但它在细胞中可能抑制了正常DNA的修复机制,使其有诱变增强作用。考虑到PCR的广泛使用,其安全性应该得到足够重视。
应用[8][9]
qPCR Master Mix可用于qPCR检测实验:
基于CTV基因组同源性较高的3′端设计筛选引物,运用SYBR GreenⅠ荧光染料法建立一种检测CTV的实时荧光RT-qPCR(Reverse Transcription and quantitative PolymeraseChain Reaction)体系,并应用于检测田间不同致病力的CTV株系。同时运用该方法建立了针对CTV强毒株CT14和弱毒株CT11的MSCP防效检测体系,对25头和50头蚜虫接种强毒分离株后的拮抗效果进行研究。
主要研究结果如下:1.在国内首次建立了检测CTV的实时荧光RT-qPCR体系,该体系具有污染几率小(全封闭体系反应)、快速(整个过程只需要2.5h)、操作和实验设计简单、成本较低等优点。2.该体系特异性强(对碎叶病、温州蜜柑萎缩病和鳞皮病无特异性扩增),线性范围宽(8×10~2~8×10~8拷贝/μL,r~2=1.000),灵敏性高(最低检测限为8×10~0贝/μL,比常规PCR灵敏100倍),重复性好(批内CV值1.6%,批间CV值3.2%),是CTV定量分析的有效手段。3.运用该体系对田间样品的检测结果表明该体系能稳定检测到田间受不同致病力感染的CTV株系,对田间的攻毒检测有指导意义。
柑橘衰退病是由柑橘衰退病毒(Citrus tristeza virus,CTV)引起的一种具有经济重要性的柑橘病毒病害,广泛分布于世界各柑橘产区。我国是柑橘生产大国,种植面积居世界,产量居世界第二。随着20世纪80年代后期我国进行柑橘产业结构调整,加大了柚类和甜橙的种植比例,CTV茎陷点型毒株对柚类和某些甜橙的危害变得日益严重。
国外的防治经验表明,在CTV强毒株和强传媒橘蚜并存的地区,弱毒株的交叉保护(MSCP,Mild Strain Cross Protection)是最有效的防治方法。建立一种快速、准确的定量检测CTV的方法不仅对预测CTV的发生和流行具有现实意义,而且对田间不同CTV株系侵染力的鉴定具有指导意义,同时运用该方法有助于对MSCP防治机理的深入研究。
参考文献
[1]Polymorphism of a specific region in gene p23 of Citrus tristeza virus allows discrimination between mild and severe isolates[J].A.Sambade,C.López,L.Rubio,R.Flores,J.Guerri,P.Moreno.Archives of Virology.2003(12)
[2]Mutational Analysis of the Replication Signals in the 3′-Nontranslated Region of Citrus Tristeza Virus[J].Virology.2002(1)
[3]Comparison of viral RNA populations of pathogenically distinct isolates of Citrus tristeza virus?:application to monitoring cross‐protection[J].A.Sambade,L.Rubio,S.M.Garnsey,N.Costa,G.W.Müller,M.Peyrou,J.Guerri,P.Moreno.Plant Pathology.2002(3)
[4]Nucleotide Sequence of Citrus Tristeza Virus Seedling Yellows Isolate[J].Cede SUASTIKA,Tomohide NATSUAKI,Hirotsugu TERUI,Takeshi KANO,Hiroyuki IEKI,Seiichi OKUDA.Journal of General Plant Pathology.2001(1)
[5]Molecular Characterization of an Isolate of Citrus Tristeza Virus that Causes Severe Symptoms in Sweet Orange[J].Zhong-Nan Yang,Deborah M.Mathews,J.Allan Dodds,T.Erik Mirkov.Virus Genes.1999(2)
[6]Complete Sequence of the Citrus Tristeza Virus RNA Genome[J].A.V.Karasev,V.P.Boyko,S.Gowda,O.V.Nikolaeva,M.E.Hilf,E.V.Koonin,C.L.Niblett,K.Cline,D.J.Gumpf,R.F.Lee,S.M.Garnsey,D.J.Lewandowski,W.O.Dawson.Virology.1995(2)
[7]The Continuous Challenge of Citrus Tristeza Virus Control[J].M Bar-Joseph,R Marcus,R F Lee.Annual Review of Phytopathology.1989
[8]M II-16,an artificial symptomless mutant of tobacco mosaic virus for seedling inoculation of tomato crops[J].A.Th.B.Rast.Netherlands Journal of Plant Pathology.1972(3)
[9]于庆涛.检测CTV的实时荧光RT-qPCR技术研发与应用[D].西南大学,2008.
